文章目錄
本篇內容包括:
- 計算各系平均分數
- R語言ANOVA單因子變異數分析案例
- R語言ANOVA單因子變異數分析-多重比較(Post Hoc)
- 應用plot以圖像來呈現ANOVA信賴區間
接著我們進入最常出現在論文中使用的方法,檢定三個以上的獨立母體平均數是不是一樣,使用變異數分析(ANOVA);透過底下的案例,可以更清楚知道如何將問題透過R語言實作出統計分析的結果。
案例:台積大學想要了解不同科系之平均英文成績是否有差異。
H0:各科系英文平均分數皆相等(μ1= μ2= μ3 = μ4 = μ5)
H1:各科系英文平均分數不全相等
將自變數中的值轉換為因子
ANOVA中的自變數形態都必須是因子,因此先將科系中的代碼轉換為因子。
collegedata$科系 <- factor(collegedata$科系,levels = c(1,2,3,4,5), labels = c("中文系","統計系","文創系","餐飲系","資管系"))
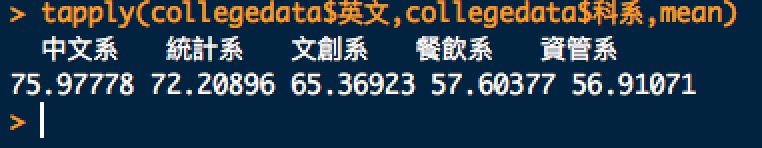
計算各系平均分數
函數:tapply(X, INDEX, FUN = NULL, …, default = NA, simplify = TRUE)
tapply(collegedata$英文,collegedata$科系,mean)
ANOVA單因子變異數分析
函數:aov(formula, data = NULL, projections = FALSE, qr = TRUE, contrasts = NULL, …)
aov(collegedata$英文 ~ collegedata$科系)
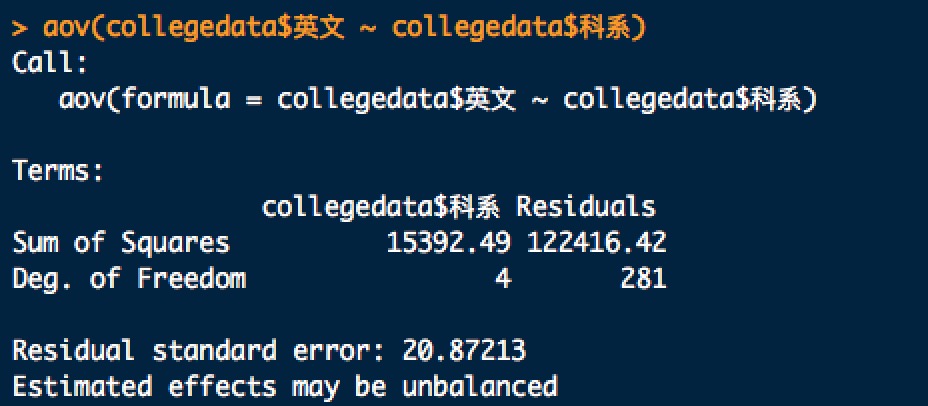
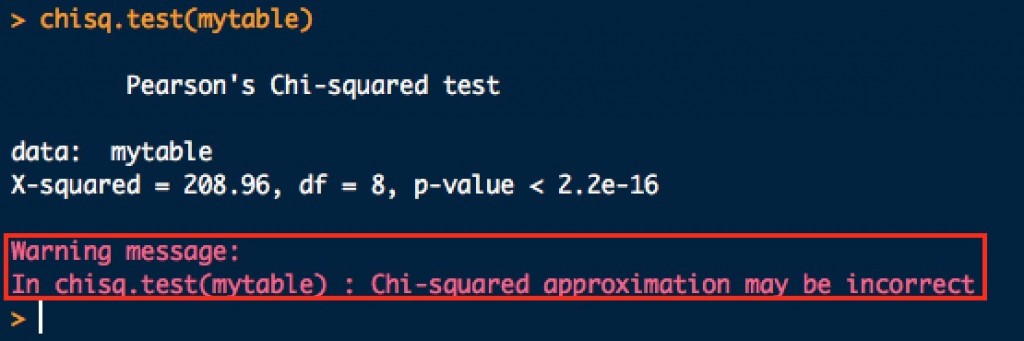
直接用aov()函數,取得的是組間(科系之間)與組內(科系內值與值之間)的平方和。因此需要再透過summary()幫我們算出 p-value 。
fit <- aov(collegedata$英文 ~ collegedata$科系)
summary(fit)
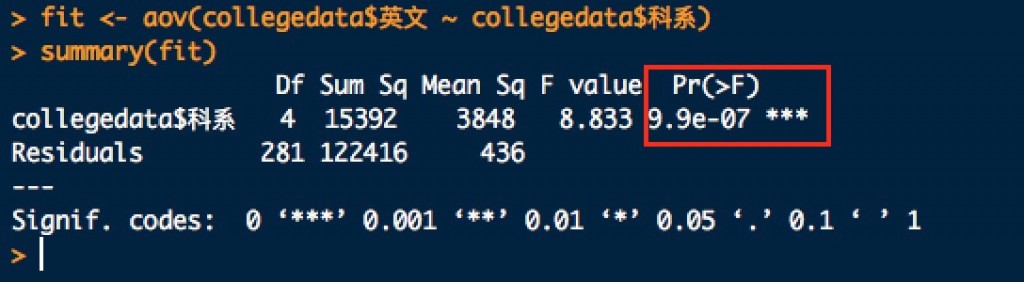
使用summary函數,取得 p-value = 0.00000099 < 0.05 ,表示各科系英文平均分數不全相等。
ANOVA單因子變異數分析-(Post Hoc)
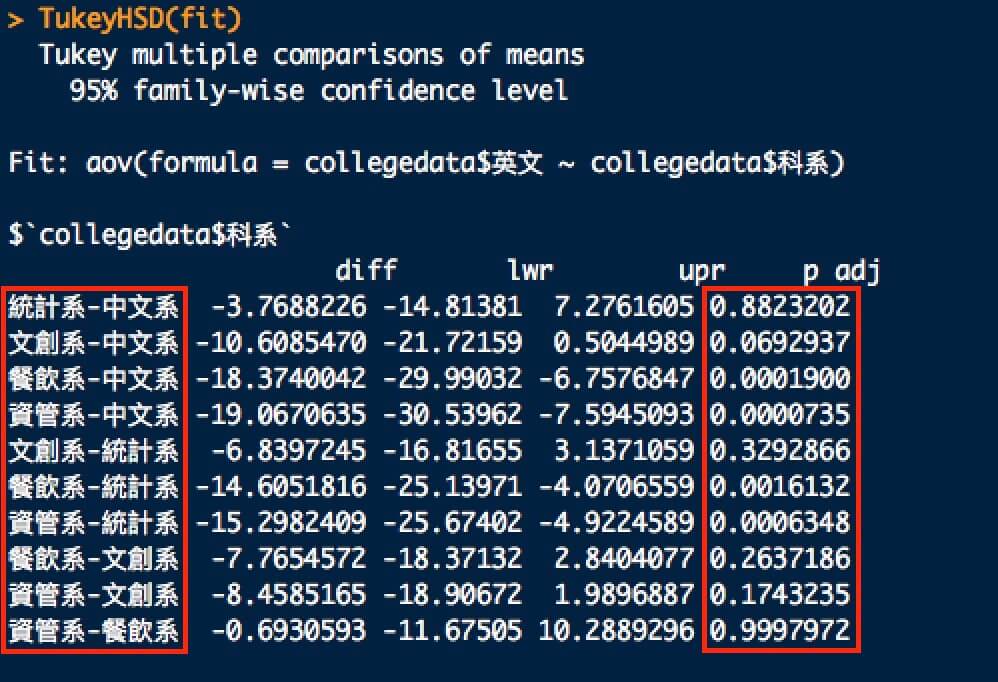
接著如果想進一步知道系與系之間相互影響的差異情況,就需要用多重比較的TukeyHSD。
函數:TukeyHSD(x, which, ordered = FALSE, conf.level = 0.95, …)
TukeyHSD(fit)

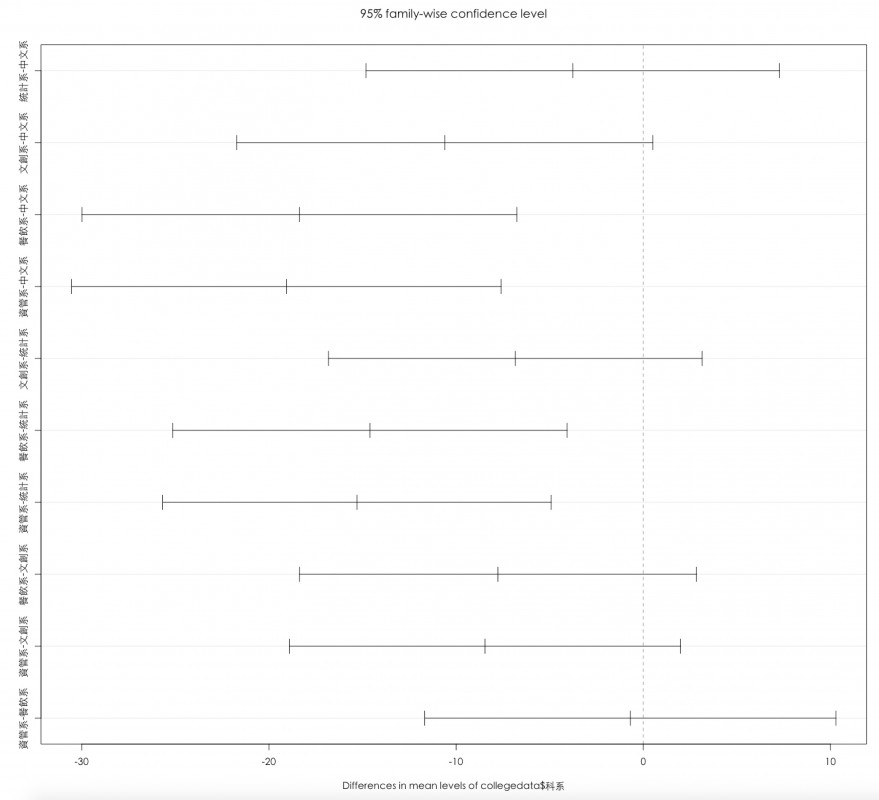
這張多重比較圖是不是覺得似乎跟SPSS跑出來的不太一樣呢?因為R語言中的TukeyHSD把重覆的都刪掉了,所以看到的是不重覆的,而且數量剛好只有一半。
有看出結論了嗎?哪幾個系與系之間無顯著差異呢?
用圖像來呈現ANOVA
因為TukeyHSD有幫我們計算出信賴區間的上下邊界,所以我們可以使用plot(),將圖畫出來。
函數:plot(x, y, …)
plot(TukeyHSD((fit), conf.level = 0.95))